Saúde
Estudo revela novo processo de desregulação do RNA que contribui para a neurodegeneração
Estudos recentes de neurociência delinearam consistentemente o papel do gene C9ORF72 no desenvolvimento de algumas doenças neurodegenerativas. Esses estudos descobriram que as mutações desse gene aumentam o risco de desenvolver esclerose...
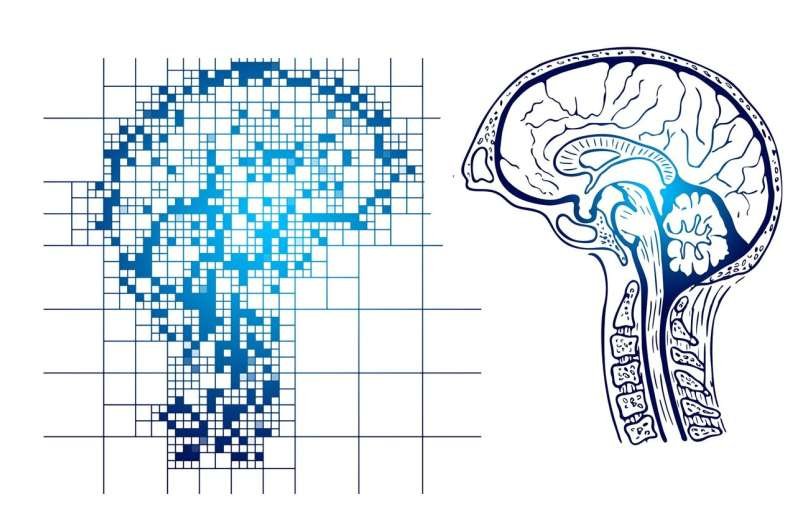
Um modelo esquemático mostrando como a hipometilação global de m 6 A em C9ORF72-ALS/FTD prejudica a função neuronal e resulta em morte neuronal ao estabilizar o RNA de repetição C9ORF72 e desregular a expressão do gene neuronal. Crédito: Nature Neuroscience (2023). DOI: 10.1038/s41593-023-01374-9
Estudos recentes de neurociência delinearam consistentemente o papel do gene C9ORF72 no desenvolvimento de algumas doenças neurodegenerativas. Esses estudos descobriram que as mutações desse gene aumentam o risco de desenvolver esclerose lateral amiotrófica (ALS) e demência frontotemporal (FTD), dois distúrbios neurodegenerativos caracterizados por deficiências motoras, problemas de comunicação e outros sintomas distintos.
Pesquisadores da Escola de Medicina da Universidade Johns Hopkins, da Universidade de Chicago, do Howard Hughes Medical Institute e de outros institutos nos Estados Unidos realizaram recentemente um estudo com o objetivo de entender melhor os processos pelos quais a mutação do gene C9ORF72 pode contribuir para o desenvolvimento de ELA e FTD. Suas descobertas, publicadas na Nature Neuroscience , destacam um caminho promissor que poderia melhorar as intervenções terapêuticas para esses distúrbios neurodegenerativos complexos.
"Meu grupo de pesquisa tem se interessado em identificar modificadores do metabolismo de repetição do RNA C9ORF72", disse o Dr. Shuying Sun, um dos pesquisadores que realizou o estudo, ao Medical Xpress. "Uma expansão de repetição de hexanucleotídeo no gene C9ORF72 é a causa genética mais prevalente de ALS e FTD. Como a expansão está localizada na região não codificadora, acredita-se que o RNA contendo repetições seja um fator importante que impulsiona a patogênese da doença."
Sun e seus colegas têm tentado identificar modificadores genéticos da produção não convencional de proteínas tóxicas poli-dipeptídeos do RNA repetido. Isso, por sua vez, poderia permitir que eles identificassem estratégias viáveis ??para prevenir o acúmulo dessas proteínas tóxicas e, assim, reduzir seus efeitos tóxicos. Em um de seus estudos anteriores, eles realizaram uma tela CRISPR-Cas9 em todo o genoma para procurar modificadores genéticos da produção de dipeptídeos.
"Descobrimos que os dois m 6 A metiltransferases são genes candidatos que podem modular o nível de poli-dipeptídeo", explicou Sun. "Também analisamos dados proteômicos públicos e descobrimos que as duas metiltransferases são reguladas negativamente em neurônios iPS de pacientes C9ORF72-ALS/FTD. Ao todo, isso iniciou nosso estudo adicional sobre a desregulação m 6 A e como isso contribui para a neurodegeneração em C9ORF72-ALS/FTD."
Sun e seus colegas realizaram uma série de análises em células -tronco pluripotentes induzidas derivadas de pacientes (iPSC) - neurônios diferenciados (iPSNs) e tecidos pós-morte extraídos de pacientes falecidos com ALS/FTD e com mutações no gene C9ORF72. Usando várias técnicas de sequenciamento genético de alto rendimento, eles avaliaram especificamente uma modificação de RNA conhecida como M 6 A (metiladenosina) e seu impacto na regulação de genes em toda a gama de RNA mensageiro expresso (mRNA), conhecido como transcriptoma.
"Também examinamos o mecanismo molecular da regulação m 6 A no RNA de repetição C9", disse Sun. "Finalmente, medimos a sobrevivência dos neurônios para examinar a eficácia do resgate modulando a via m 6 A. Nossas descobertas revelam uma nova camada de regulação do RNA que desempenha um papel crítico no mecanismo patogênico subjacente à neurodegeneração."
No geral, as descobertas reunidas por esta equipe de pesquisa mostraram que a modificação m 6 A na sequência genética C9ORF72 de repetições expandidas facilitou o decaimento do RNA por meio do leitor nuclear YTHDC1, regulando assim as repetições. Além disso, a redução em m 6 A pareceu facilitar o acúmulo de RNAs repetidos e a codificação de proteínas tóxicas conhecidas por estarem associadas à neurodegeneração.
Ao elevar a metilação do m 6 A, Sun e seus colegas foram capazes de reduzir significativamente o nível de RNAs repetidos, protegendo os neurônios em que estavam experimentando e reduzindo o risco de decaimento. No futuro, esses resultados podem abrir caminho para o desenvolvimento de tratamentos mais eficazes para ALS e FTD, por exemplo, levando a estudos avaliando o valor da terapia epitranscriptômica como uma intervenção potencial.
"Gostaríamos de testar ainda mais a eficácia do resgate in vivo usando modelos de camundongos apropriados", acrescentou Sun. "Também estamos interessados ??em desenvolver abordagens farmacêuticas direcionadas a essa via. Além disso, esperamos investigar os mecanismos subjacentes da doença envolvendo o metabolismo do RNA e a desregulação do m 6 A na ELA esporádica e em outros distúrbios neurológicos".
Mais informações: Yini Li et al, N6-metiladenosina (m6A) globalmente reduzida em C9ORF72-ALS/FTD desregula o metabolismo do RNA e contribui para a neurodegeneração, Nature Neuroscience (2023). DOI: 10.1038/s41593-023-01374-9
Informações da revista: Nature Neuroscience